写在前面的
昨天大概理解了基于Bisulfite测序的甲基化数据分析背景。今天马上就读到卢煜明院士团队在2019年于《Genome Research》期刊发表的利用重亚硫酸盐测序的方法开发了准确鉴定5’锯齿状末端cfDNA的Jag-seq测序方法。Jag-seq测序方法的建立将cfDNA生物信息的研究维度从基因组/表观组提升至片段组(fragmentomes)
锯齿状cfDNA
前面的推送提到,cfDNA作为生物标志物在肿瘤早筛、产前诊断等领域具有非常巨大的应用前景。因此关于cfDNA相关的研究一直是热点。前几天的推送提到,作为无细胞游离的DNA,cfDNA常常来自于疾病早期被清除裂解的细胞释放的核酸。因此cfDNA上通常保存了疾病早期的基因变异信息。甲基化修饰作为调控基因表达影响细胞命运介导疾病产生的重要表观遗传学现象,cfDNA片段上也常常携带着甲基化表观遗传信息。如果把cfDNA的核苷酸变异信息和甲基化信息视为在基因组和表观组两个维度的重要信息的话,那么卢煜明院士团队在2019年发表的这篇论文将cfDNA的信息维度再一次突破至片段维度,写到这里可能读者还有些陌生,如果你将突变或者甲基化抽象为物理学中的质点,那么cfDNA本身的片段特征则是一条线,该团队的科学家在点的基础上运用了相对更宏大的视角从更高的领域在观察cfDNA。在该团队前期的研究中发现cfDNA片段的产生并非随机的,比如上上篇推送报道了该团队曾发现cfDNA片段中的序列特征可以追溯其组织来源,因此该团队认为cfDNA本身的片段整体特征或许还蕴含了更多的信息。在这个背景下,cfDNA的片段物理特征这一概念被提出。由于cfDNA来通常来自于细胞的主动或被动裂解,DNA片段也常被DNase等相关酶切割。实际的cfDNA完整性如何还不清楚,特别是cfDNA是否存在不完整的双链?例如在双链部分会存在额外延伸的单链?(该团队将这种需要被探索的cfDNA取了很形象的名字,锯齿状cfDNA)这些片段特征需要被进一步探索。但是普通的高通量测序会使用DNA连接酶补齐末端。这就导致不能直接通过高通量测序完成对这类cfDNA的探索。因此需要开发新的测序技术实现精准探索是否存在锯齿状cfDNA。
Jag-seq测序方法鉴定锯齿状cfDNA
该团队根据cfDNA本身的特点及团队前期的积累首先构思了第一种检测双链cfDNA分子5’锯齿状末端的方法。首先利用DNA聚合酶将非甲基化的dNTPs连接到这类cfDNA分子末端以补齐双链DNA。由于这类cfDNA分子内部存在高度甲基化的C(黑色点),而新加入的C是非甲基化的(白色点),因此在重亚硫酸盐(Bisulfite)处理下未甲基化的C全部变成了T,甲基化的C仍然保留。由于cfDNA分子长度一般为166bp,大部分cfDNA分子长度介于156bp到176bp之间。在5’锯齿状末端类型的cfDNA分子中,双端NGS测序Forward片段(R1)将覆盖大部分原始cfDNA区间,Reverse片段(R2)除了覆盖部分原始cfDNA区间,还覆盖了后续填补的非甲基化区间。那么R2的reads一定存在一部分低甲基化水平的区间。如果以cfDNA分子作为参考,Read1甲基化程度一定很高,而Read2的甲基化程度将沿着cfDNA分子5’>3’方向降低。(因为填充的末端是未甲基化的)。
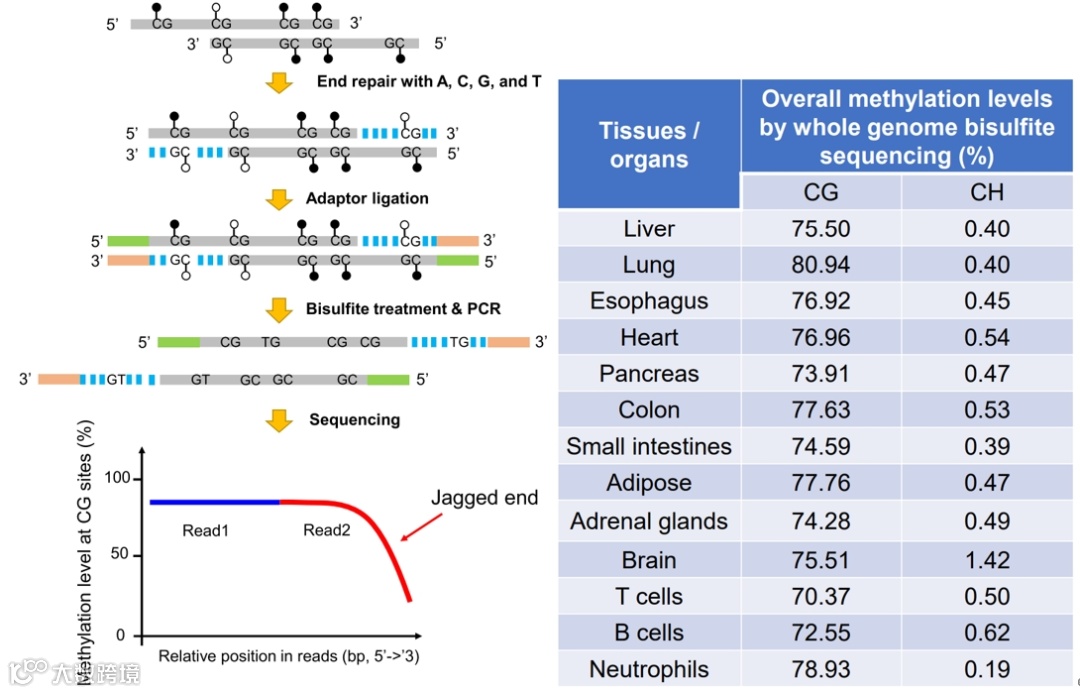
看完这种构思我陷入了疑惑,为什么cfDNA分子一定是高度甲基化的,随后我在该团队发表的补充材料中发现了支撑他们产生这种想法的重要结果,在上图的表格部分是该团队分析整理的机体组织不同位点上CG上下文序列和CH上下文序列中C的平均甲基化水平,其中H代表简并碱基(A/C/T)。这个表格揭示了当与G在一起的C碱基的甲基化可能性远远大于与A或C或T碱基在一起的C碱基的甲基化可能性。因此对于这类双链cfDNA分子中,原始DNA分子内的CG序列的C大部分是甲基化的。而后续由非甲基化dNTPs填补的平末端序列才能与原始cfDNA序列的甲基化水平产生显著的差异。不仅如此,作者还提出了一个公式用于量化cfDNA锯齿状指数的公式,分别用M1和M2代表Read1和Read2的甲基化密度,由于Read1的甲基化水平一定高于Read2,所以百分化的JI-U值一定在0~100区间内。
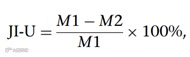
作者首先将他们已发布的10份血液细胞DNA样本和30份血浆DNA样本进行了测试,对血液细胞进行超声处理后与血浆中cfDNA分子进行对比。果然发现了5’锯齿状cfDNA存在。并且血浆中cfDNA的锯齿化程度较高。

由于上面检测策略仅仅针对富含CpG二核苷酸序列的cfDNA,相比与CG序列,CH序列在基因组中覆盖得更多,为了提高对富含CH序列的cfDNA锯齿状末端的检测能力。该团队在方法一的基础上构思出了方法二。如下图所示,由于CH序列的C通常情况是非甲基化的,那么如果引入的dNTPs中dCTPs是甲基化的那么就能引入高甲基化水平的修复末端。在重亚硫酸盐处理测序后,Read1覆盖了全部低甲基化水平的原始cfDNA片段所在区间,Read2除了覆盖原始cfDNA片段所在区间还覆盖了高度甲基化的修复末端。那么此时甲基化水平的曲线和方法一相反。不仅如此,该团队还注意到这种情况中有一类特殊的cfDNA分子还可能具备被解析出精确锯齿状末端的长度。如果cfDNA的3’末端是未甲基化的C,而引入甲基化的C后再BS-seq测序数据中会产生TC分子标记,通过BS-reads-alignment一定会找到精确的TC(read)-map-CC(genome)的位点。此时锯齿状末端的长度定量成为了可能。作者将这种特殊的可定量锯齿状cfDNA锯齿末端长度的情况以其分子标记命名CC-tag命名,第一个C指代cfDNA原始的3’末端未甲基化的C,第二个C指代添加的甲基化C。
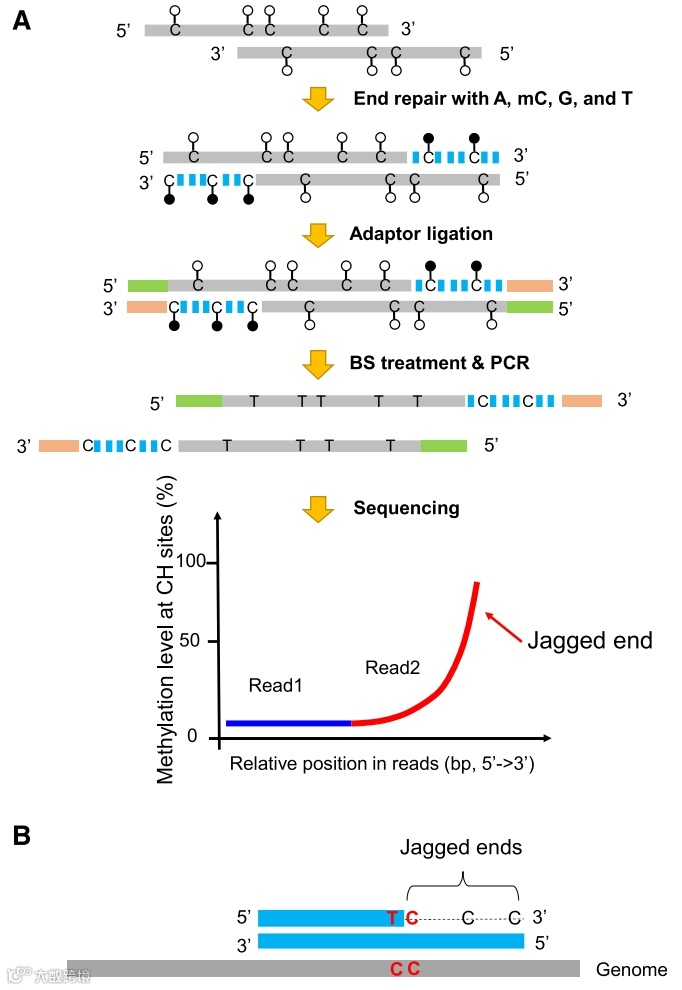
在这一步方法构建完成后,作者并没有忙着马上对实际样本进行应用测试,而是先人工合成了一段13nt和22nt锯齿状末端的cfDNA,用于评估甲基化的胞嘧啶是否可以正常添入修复末端。我其实不是很确定作者他们为什么要这样做,可能是自己的分子生物学背景不扎实。我猜想有两种可能(不是特别确定,还望读者们指点):1.甲基化的dCTP可能不容易被DNA聚合酶和连接酶体外加入延伸的DNA链中,可能会影响方法二的正常进行。2.甲基化的dCTP是否可能影响cfDNA链中的CH位点的未甲基化胞嘧啶?如果可能影响,那么方法二不能按照期望根据甲基化信号推断cfDNA的锯齿化程度。因此有必要在分析实际样本前排除这两种可能性。(这是我自己不扎实的分子生物学知识引导我猜测的,如有错误还望读者私信指出,感谢)。
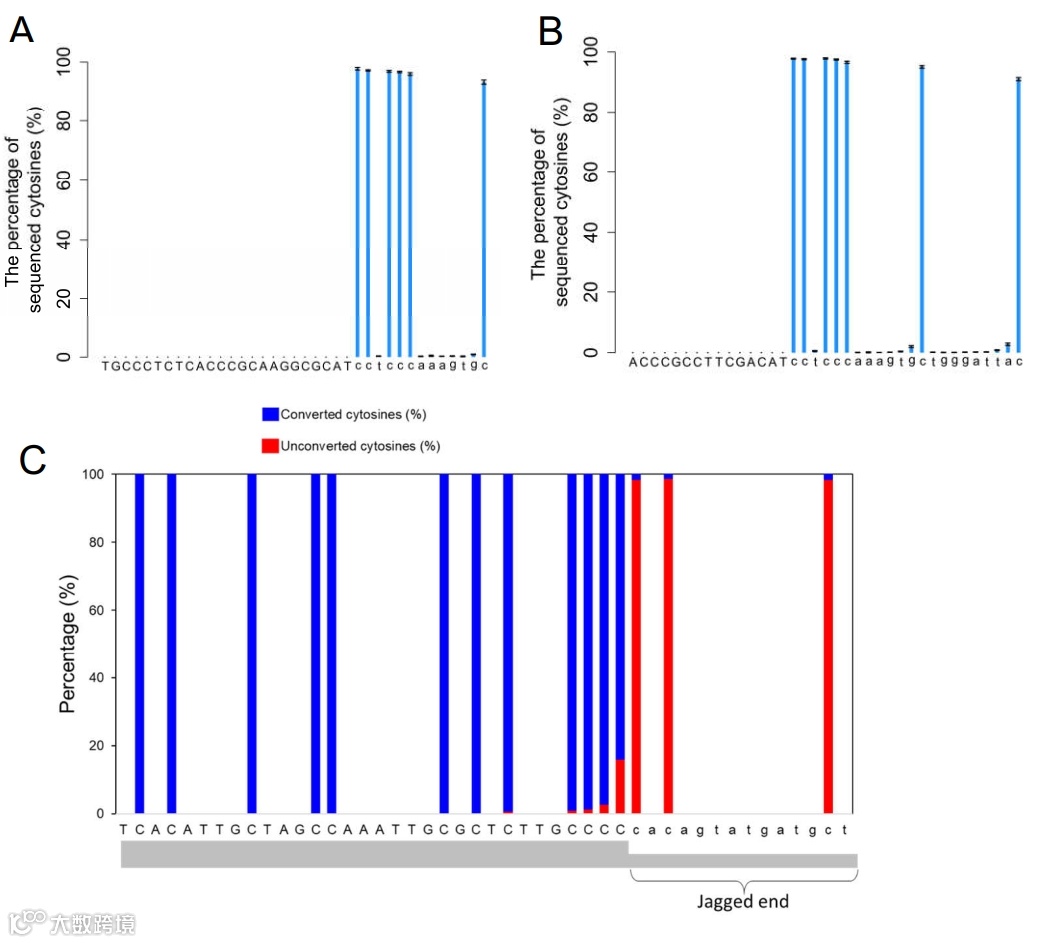
紧接着作者评估了甲基化的胞嘧啶能否可以正常添入,如下图A所示,由于锯齿状末端是人为合成的,长度确定(13nt),因此如果甲基化的胞嘧啶可以正常添入,那么最后13nt能在重亚硫酸盐测序数据中被直接捕捉,事实上这13nt的确被测到了,且C位点的甲基化信号极强。另一方面,这13nt上游的C位点均是CH位点,是非甲基化位点,测序数据中这些C位点的甲基化占比几乎都为0,说明原始cfDNA中CH位点的非甲基化胞嘧啶均在Bisulfite环境中有效地被转化为了T。另外作者还用22nt的锯齿状末端评估了CC-tag标签确实可以准确定量对应的特殊型cfDNA锯齿状末端长度。
当完成上述两种方法构建和验证后,作者团队将这些方法应用于评估胎儿和母体、肿瘤来源和非肿瘤来源等样本cfDNA锯齿化情况。团队的研究结果提示胎儿cfDNA的锯齿末端平均长度比母体更长,肿瘤来源的cfDNA呈现更多的锯齿末端。此外研究同样发现锯齿状末端大小本身可能受到cfDNA片段大小及核小体模式的影响。
当我了解完作者的研究后我产生了一个疑问,目前研究建立了解析5’锯齿化末端的cfDNA锯齿化情况的方法,3’锯齿化末端的cfDNA如何测定?我猜测因为DNA聚合酶是从5’>3’方向合成DNA,5’突出的锯齿化末端的3’部分可以很容易得被DNA聚合酶以互补链为参考添加末端序列修复,但是3’端突出的锯齿化末端,很难通过DNA聚合酶逆着方向补齐5’端。如果要完成3’锯齿化末端的检测,可能需要更多基因工程或分子生物学探索。
参考
Jiang P, Xie T, Ding SC, Zhou Z, Cheng SH, Chan RWY, Lee WS, Peng W, Wong J, Wong VWS, Chan HLY, Chan SL, Poon LCY, Leung TY, Chan KCA, Chiu RWK, Lo YMD. Detection and characterization of jagged ends of double-stranded DNA in plasma. Genome Res. 2020 Aug;30(8):1144-1153. doi: 10.1101/gr.261396.120. Epub 2020 Aug 14. PMID: 32801148; PMCID: PMC7462074.
写在后面的
从前期的科学积淀到开发出新的测序技术研究更高维度的cfDNA片段组学特征,这是一个非常巨大的科学突破。


