文献信息:

作者:Heleen T. Ouboter, Rob Mesman, Tom Sleutels, Jelle Postma, Martijn Wissink, Mike S. M. Jetten, Annemiek Ter Heijne, Tom Berben, Cornelia U. Welte
发表时间:17 February 2024
https://doi.org/10.1038/s41467-024-45758-2
Nature Communications 影响因子:16.6
背景介绍
厌氧甲烷氧化(ANME)古菌是一类尚未培养的环境关键微生物,可氧化强效温室气体甲烷,是调控温室效应的“隐形阀门”,却至今未被纯培养。它们能把甲烷电子“递”给伴生菌、铁锰矿物或人工电极,这条胞外电子传递(EET)路径被视为微生物圈的“暗线”,机制长期空白。
最新研究在生物电化学系统中定向富集淡水类群 ANME-2d“Ca. Methanoperedens”,仅3周就让其占阳极生物膜 82%,甲烷氧化贡献 91–93% 的电流;多电位测试显示,EET 策略几乎不变,核心可能是一套尚未命名的短程电子蛋白复合体,以及类似OmcZ 纳米导线。该结果首次把 ANME 的“电子路线图”拉到可观测尺度,为解锁剩余 90% 未知甲烷汇提供跳板。
图文解读

图1 | 三项实验流程总览
图中标出实验过程中的培养基更换、甲烷依赖性电流测试、循环伏安(CV)与极化扫描、活性测试,以及宏基因组与宏转录组采样和显微观察等事件。实验1所有体系均设生物学三重复;转录组RNA样本采集:0 mV三重复(2生物+1技术,n=2)、200 mV三重复(3生物,n=3)、400 mV四重复(3生物+1技术,n=3)、600 mV四重复(3生物+1技术,n=3),硝酸盐生长条件三重复(3生物,n=3)。转录组RNA样本数差异源于古菌生物量低且RNA提取困难。宏基因组DNA提取:实验1每种条件取1个生物阳极,实验2进行1次。

图2
实验1获得的生物电化学数据,包括不同电位下的测量结果。图中显示三次生物学重复的平均值(n = 3)。
A 更换培养基后,所有系统初始在0 mV vs SHE下运行,随后切换至不同电位(0 mV、200 mV、400 mV或600 mV vs SHE)时的电流产生情况;
B 用氩气替换甲烷以测试甲烷依赖性电流;
C 循环伏安扫描;
D 极化扫描,虚线表示CH₄/CO₂氧化还原对的氧化还原电位−0.249 V。绿色:0 V vs SHE条件;粉色:200 mV vs SHE条件;紫色:400 mV vs SHE条件;橙色:600 mV vs SHE条件。源数据作为源数据文件提供。

图3 | 培养基更换次数与孵育时间对甲烷依赖性电流百分比及“Ca. Methanoperedens”相对丰度的影响
图中蓝色圆点表示微生物群落中“Ca. Methanoperedens”的相对丰度(%),红色圆点表示甲烷依赖性电流百分比,两者均相对于培养基更换次数绘制。宏基因组丰度数据每种条件采集一次生物学重复(n = 1)。甲烷依赖性电流数据:“无更换”条件采集一次生物学重复(n = 1),更换一次培养基采集五次生物学重复(n = 5),更换两次培养基采集四次生物学重复(n = 4)。甲烷依赖性电流平均值以水平线标示。源数据作为源数据文件提供。

图4 | 使用不同显微技术观察生物膜
A–I 共聚焦激光扫描显微镜结合荧光原位杂交(FISH):FLUOS探针(青)靶向“Ca. Methanoperedens”,Cy5探针(黄)靶向细菌,Cy3探针(绿)靶向Geobacter sp.
J 扫描电镜(SEM):距电极网较远处可见“Ca. Methanoperedens”大簇,金网附近为致密杆状细菌生物膜
K, L 透射电镜(TEM):K显示距金网较远的“Ca. Methanoperedens”细胞,L显示金网附近的杆状细菌细胞
本图全部样品来自实验3;A–H三次生物学重复(n=3),C、F、I两次生物学重复(n=2);SEM两次生物学重复(n=2),TEM两次生物学重复(n=2),均观察到相似结果

图5 | 微生物群落的宏基因组与宏转录组分析
A 宏基因组reads和B RNA-seq reads分别映射到宏基因组组装基因组(MAGs)后,在科水平获得的相对丰度与相对活性。Methanoperedenaceae与Geobacteraceae各含一个MAG,分别被GTDB鉴定为"Ca. Methanoperedens"与Geobacter sp.。仅展示相对丰度或活性>2.5%的分类群。RNA样本采集:0 mV三重复(2生物+1技术,n=2);200 mV三重复(3生物,n=3);400 mV四重复(3生物+1技术,n=3);600 mV四重复(3生物+1技术,n=3);硝酸盐生长条件三重复(3生物,n=3)。转录组RNA样本数差异源于古菌生物量低且RNA提取困难。宏基因组DNA提取:实验1每种条件取1个生物阳极,实验2进行1次。源数据作为源数据文件提供。
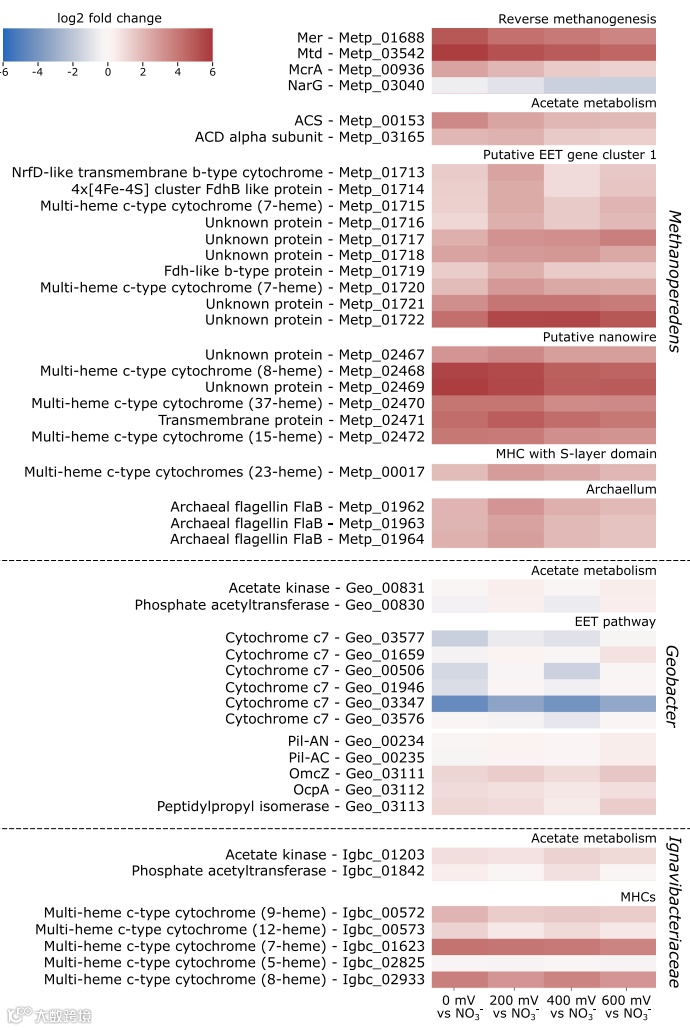
图6 | 电极条件(0 mV、200 mV、400 mV、600 mV)与硝酸盐条件的差异基因表达分析
图中展示“Ca. Methanoperedens”两个可能参与EET的基因簇,以及Geobacter sp. EET相关基因;同时列出电极条件下归一化计数≥30的Ignavibacteriaceae多血红素细胞色素(MHCs),因其EET机制尚不清楚。特别提及Geobacter与Ignavibacteriaceae是因为它们在本研究中被证实具有活性。此外,还加入这三种微生物参与乙酸代谢的酶基因,因为它们可能在“Ca. Methanoperedens”与这两种细菌间的互作中发挥作用。源数据作为源数据文件提供。
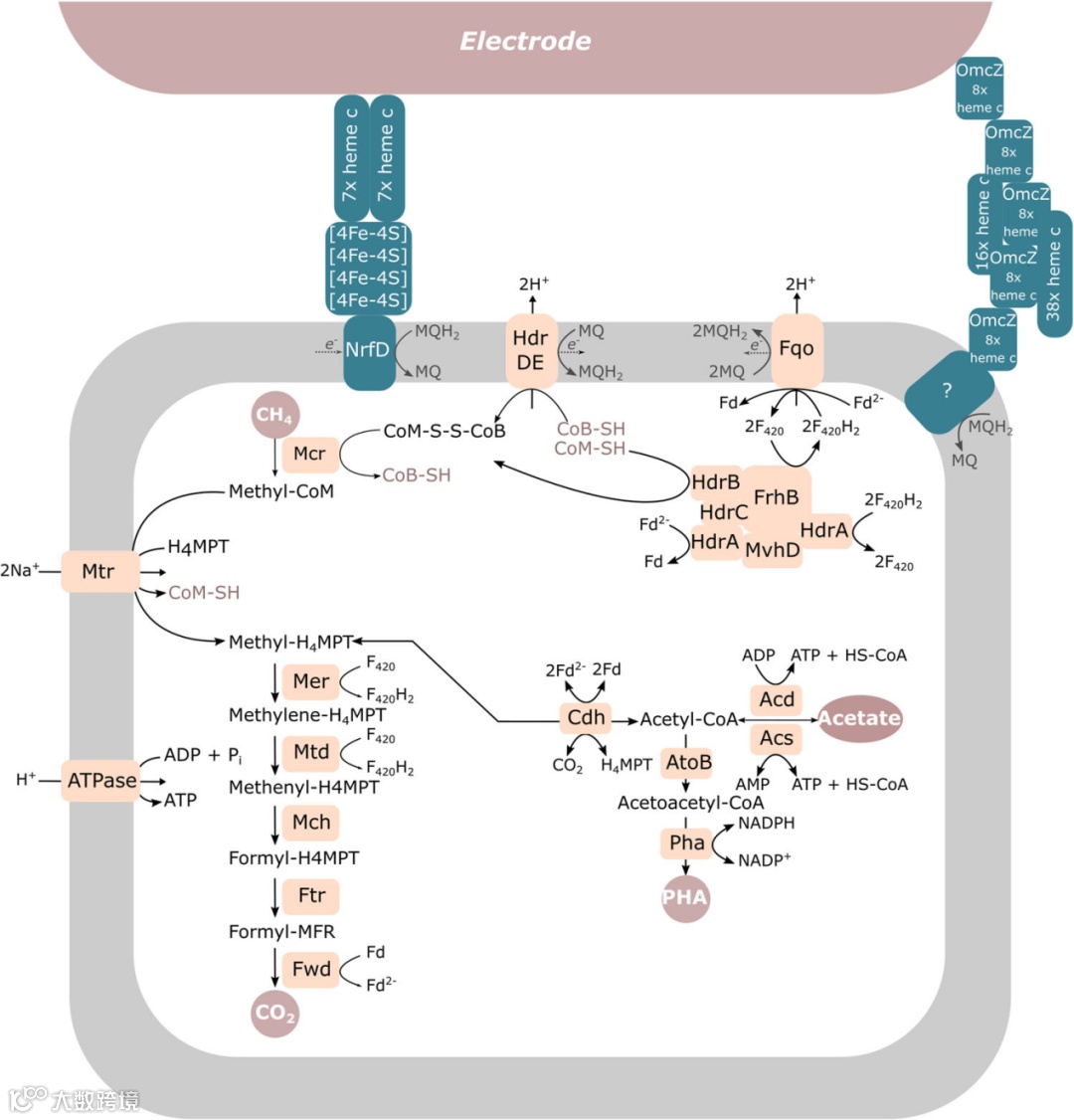
图7 | “Ca. Methanoperedens” proposed 代谢及其向电极的假定EET机制总览
合成生物学相关内容讨论与合作,及文章免费投稿
欢迎联系我:shalafangjian
了解更多合成生物学内容,请关注 iSynFox 🦊:


