DNA language model GROVER learns sequence context in the human genome
DNA语言模型GROVER学习人类基因组中的序列上下文

📑 基本信息
- 期刊
: Nature Machine Intelligence - 时间
: 2024-07-23 - DOI
: 10.1038/s42256-024-00872-0 - 作者
: Melissa Sanabria, Jonas Hirsch, Pierre M. Joubert, Anna R. Poetsch* - 机构
: Technische Universität Dresden, Center for Advanced Systems Understanding (CASUS), Helmholtz Zentrum Dresden-Rossendorf (HZDR), National Center for Tumor Diseases (NCT)
🔍 导读
该研究开发了基于字节对编码的DNA语言模型GROVER,通过自定义的next-k-mer预测任务优化词汇表,成功学习了人类基因组的序列上下文和语言规则。GROVER不仅在基因组生物学任务中表现优异,还能从序列关系中学习功能基因组学注释,为解读"生命密码"的语法规则提供了新工具。
🧩 引言
背景与意义
人类基因组草图已问世20多年,但对"遗传密码"的理解仍然有限。基因组中仅1-2%的DNA三联体编码氨基酸,其余部分包含基因调控、转录结构、复制和稳定性等多层次编码信息。大语言模型的兴起为全面提取这些复杂编码层次提供了前所未有的机会。

图表解读:该图展示了DNA字节对编码和BERT架构的基本原理,说明了如何将基因组序列转化为可供语言模型学习的token,体现了将自然语言处理技术应用于基因组学的创新思路。
现有不足
现有DNA语言模型如DNABERT和Nucleotide Transformer主要使用固定长度的k-mer作为词汇表,存在频率分布不均衡的问题。人类基因组中k-mer频率差异可达1000倍,这种不平衡会导致模型训练困难,可能更多学习频率特征而非真正的基因组语言上下文。
问题与动机
基因组序列虽然在结构上类似自然语言的语法、句法和语义,但缺乏明确的"单词"概念。如何为DNA序列构建最优词汇表,使语言模型能够有效学习基因组的语言规则和生物学意义,是当前面临的核心挑战。
目标与创新
研究目标是建立频率平衡的人类基因组词汇表,训练名为GROVER的基础DNA语言模型。创新点包括:采用字节对编码生成多个词汇表、通过next-k-mer预测任务选择最优词汇表、深入分析模型学习的基因组信息内容,为提取"生命密码"语法规则奠定基础。
📊 方法
数据来源与类型
使用人类基因组GRCh37(hg19)组装版本,仅包含A、C、G、T核苷酸序列。将每条染色体分割为20-510个token的窗口,其中50%概率选择510token长度,另50%随机选择20-510之间的长度。80%窗口用于训练,20%用于测试。

图表解读:该图展示了通过next-k-mer预测任务选择最优词汇表的过程,显示600轮字节对编码在多个k-mer预测任务中均表现最佳,为GROVER模型选择提供了客观依据。
核心方法与技术
采用字节对编码算法生成频率平衡的词汇表,从4个核苷酸开始,逐步合并最频繁的token对形成新token。使用BERT架构训练掩码token预测任务,模型包含12个transformer层,支持最大510token输入长度,采用交叉熵损失函数。
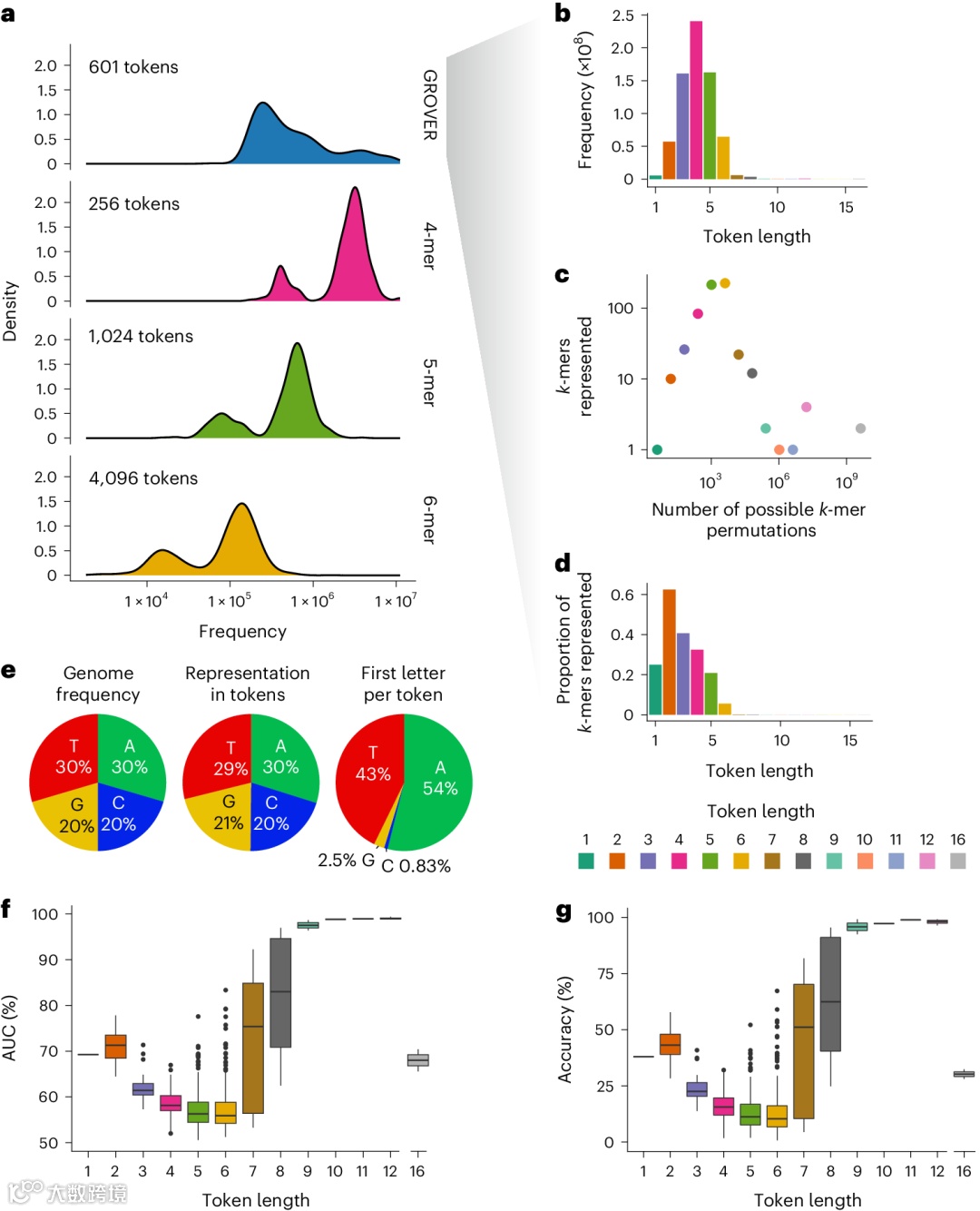
图表解读:该图展示了GROVER词汇表的频率平衡特性,显示BPE-600词汇表相比固定k-mer词汇表具有更均匀的频率分布,大部分token为4-mer,平均长度4.07,有效解决了频率不平衡问题。
研究流程
首先对人类基因组进行100-5000轮字节对编码生成多个词汇表,然后训练相应的BERT模型进行掩码token预测。通过next-k-mer预测任务评估不同词汇表的性能,选择600轮编码作为最优方案。最后分析token嵌入和上下文学习能力。
📈 结果
最优词汇表性能显著提升
GROVER在next-6-mer预测任务中达到2%的准确率,显著超越次佳模型DNABERT-2的0.6%准确率。固定k-mer BERT模型包括NT的准确率均不超过0.4%。掩码token预测任务中,GROVER达到21%准确率,允许前60个预测token时准确率提升至75%。

图表解读:该图展示了GROVER token嵌入学习的基因组信息内容,包括频率、GC含量、AG含量和token长度等特征。与静态Word2Vec嵌入相比,GROVER显示出更强的上下文学习能力。
基因组语言规则学习
GROVER成功学习了token特征和序列上下文信息。主成分分析显示,PC1强烈关联token频率(Spearman's R=0.88),PC2关联GC含量(R=-0.96),PC3关联AG含量(R=0.94)。模型还能识别重复元件、染色质状态和复制时序等生物学特征,显示出从序列关系中学习功能基因组学注释的能力。

图表解读:该图展示了GROVER学习的基因组区域上下文信息,通过UMAP可视化显示模型能够区分不同类型的重复元件、染色质特征和方向性信息,证明了模型学习生物学相关序列上下文的能力。
生物学任务优异表现
在三个代表性微调任务中,GROVER均表现出色。启动子识别任务(Prom300)中达到99.6% MCC,远超4-mer模型的79%。启动子扫描任务(PromScan)中达到63% MCC,优于NT的52%。CTCF蛋白-DNA结合预测任务中达到60% MCC,与DNABERT-2的59%相当。
💡 结论
主要贡献
创新词汇表构建方法:首次将字节对编码成功应用于人类基因组,通过next-k-mer预测任务客观选择最优词汇表,解决了固定k-mer方法的频率不平衡问题,为DNA语言模型提供了新的词汇表构建范式。
基因组语言规则深度学习:GROVER不仅学习token身份特征,还能从序列上下文中学习染色质状态、重复元件、复制时序等生物学信息,证明了基因组序列中蕴含丰富的可被语言模型捕获的信息内容,为理解"生命密码"语法规则奠定了基础。
局限性与展望
当前方法仅使用人类基因组训练,限制了训练数据规模,可能不适用于较小基因组。标准基因组生物学任务在很大程度上可通过token频率预测,表明需要开发更多独立于token频率的任务来评估真正的生物学序列上下文学习。未来可通过提取学习表征、注意力机制分析等方式,进一步挖掘基因组信息内容,推进个性化医学发展。
想做自己的垂直领域大数据研究(电池、陶瓷、金属、医药、蛋白、环境、生态、材料、农学、地质等),可使用类似方式完成定制化数据集搜集搭建,TAG-HUB也可提供定制化科研数据集挖掘服务。




