⭐ 添加小助手微信(SDbioinfo_2023)
可领取👉 国内AI4Science团队汇总表(含高校、公司和机构)
声明:此文由“AI4Science 目前是原来搞 Science 的做得更好,还是搞 AI 的做得更好-Sponge的回答-知乎”作者在其原回答基础上增添修改而来,原文链接:
https://www.zhihu.com/question/653239898/answer/1937611016646168620
“AI4Science中,Science背景和CS背景到底谁做得更好?”
这个问题近来关注颇多,在此我以在AI For Biology(AI4B)的一点浅薄经验来讨论一二。首先是我个人认为AI4B现在总体可以分为三个方向,在末尾会给出我的回答:
方向一
以Science的问题为主,AI从作为一环来解决特定问题或者在湿实验的基础上做一延申,到整体上自动化科研过程
这一方向多为 Science 出身的人在做,基本都在生物医药领域。他们针对的问题往往伴随湿实验成本高,周期长,数据量少等背景。最突出的例子应该是 AlphaFold [1],初代聚焦预测蛋白质结构这一当时的难题来构建模型,并很好的解决了此问题,AlphaFold3 [2]则延伸到了更多结构和结合问题,最终在 2024 年获得诺奖。
此方向文章的水平和质量与所研究的Science问题密切相关。举个例子,前两年预测生物分子的简单分类任务(比如判断一个序列是否为某种肽),不需要设计多么复杂的模块,仅在使用了如 ESM2 [3]之类 Foundation Model 的表征之后,从指标上来说就比之前的机器学习模型效果提升显著。这类研究也不用做湿实验验证,一般文章发在二区往上。反过来那些涉及前沿的、涉及基础理论的、应用价值大的、或亟需解决的问题的重要研究工作,其文章基本都在子刊或领域内顶刊,乃至正刊发表,往往都以湿实验为主。比如,在通过丰富的实验环环相扣解析和证明某个癌症致病机制的同时,可以借病理切片或者转录组的数据来训练一个模型对癌症做一预测或分型,又或者是某种新的测序技术,通过新数据来训练模型分析结果,并可反向证明新技术的性能。
此外,这个方向的发展现在已不限于以上提及的让 AI 作为一环参与项目,在Agent大行其道的背景下,斯坦福大学推出 Biomni [4] 科研智能体模型,实现了自然语言下命令。如“请用 scanpy 进行标准化和聚类,展示UMAP,并标注 CD3 和 CD19 表达”,即可模型自动全流程执行单细胞分析的功能。最近 Biomni 又宣布与开源框架 PyLabRobot [5]集成,其现在能从自然语言直接生成自动化实验协议,搭配自动化实验平台,从细胞接种到加药,全流程自动执行,如以下由Biomni一作Kexin Huang博士在X平台所展示视频:
AI 已经从方法或工具的一部分往替代人工,实现科研全流程自动化的方向发展。更多的,现在一些重要的细分问题上,比如药物相关的抗体设计,也有欠缺数据或高质量数据的说法。但如果通过 Agent系统通过 AI 实现湿实验获得数据,到干实验分析的自动化,那即可快速积累实验数据,反向再去训练专精模型。由此数据集的问题也就迎刃而解。当然,这部分模型还并未完全成熟,离真正的落地和做出实际成果还有距离。
总之,除去 Agent 这部分,这一方向的重点在于干湿结合,要么湿实验为主,要么有丰富的湿实验来验证模型的效果或泛化性能。想实际证明 AI 在生物学的有效性,湿实验是不可绕开的一环,指标只是参考,不能完全衡量其在现实中的性能。
方向二
以AI为主,对Science的问题或者数据进行建模,由湿实验验证效果,或通过模型解析未知的机制
这一方向多为CS出身在做,他们在发表上一般会更倾向于顶会,当然一些重要的工作还是发在了CNS上,比如 DeppSeek-R1 作为封面文章发表于Nature。
具体比如各种 Biological Foundation Model(BFM),蛋白如 ESM 系列,Genome 如 EVO1/2[6-7]、DNABert1/2[8-9]、GENErator[10]、Nucleotide transformer [11]等,RNA 如 AIDO.RNA [12]、RiNALMo [13]、 RNAErnie [14]。特别是 DeepMind 开发的 AlphaGenome [15],其以 1兆碱基(1Mb)的 DNA 序列为输入,能预测涵盖基因表达、转录起始、染色质可及性、组蛋白修饰、转录因子结合等 11 种模态的功能基因组轨迹,部分预测可达到单碱基对分辨率,展现了序列驱动模型在多模态建模上的进阶能力。
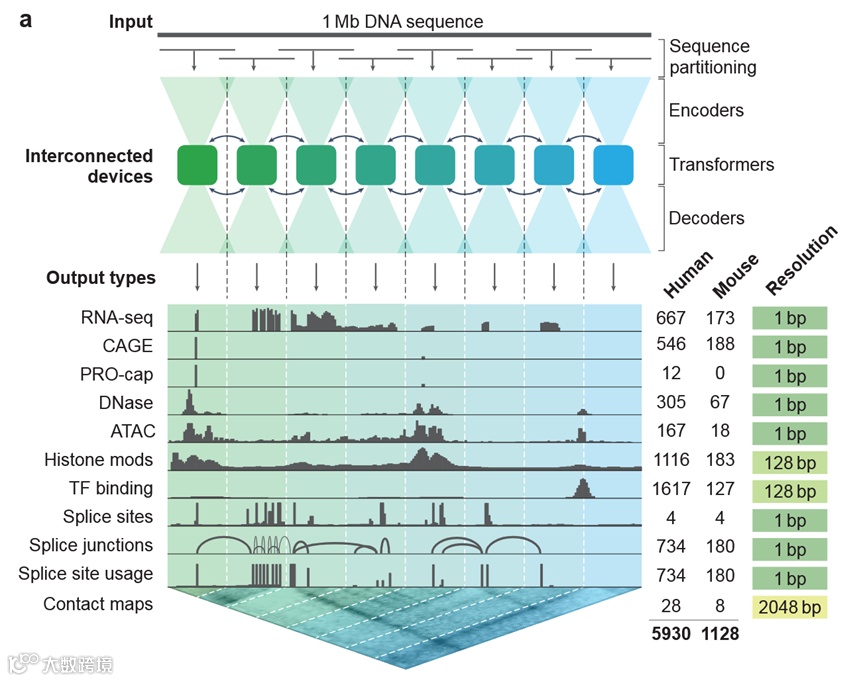
Model Overview of AlphaGenome[15]
但无论是 AlphaGenome,还是其他序列模型都暗暗遵循着一个共同前提:序列决定一切(序列中已含有生命过程中所有的信息)。然而,DNA 在一个生物体的所有体细胞中都是相同的,但体细胞却分化出了数百种细胞类型,表观遗传学揭示了从序列、表达到人体表型这一过程中间的转录调控环节还有大量复杂的机制蕴含其中,后续有一篇描述 DNA 序列数据特性的文章会对这部分进行更丰富的说明。
总之,在各种测序技术飞速发展的情况下,一个更大的趋势即越来越多的组学数据被纳入此范式之中被建模,如使用基因表达数据训练的单细胞模型如scGPT[16]、Geneformer[17]、scFoundation[18],同样有DeepMind参与的Cell2Sentence[19],以及前段时间很火的由扰动基因表达数据训练的Virtual Cell(如STATE[20])等。
目前最新的 BFM 已正式步入表观遗传领域,如清华大学最近推出的首个单细胞表观基因组基础模型 EpiAgent[21],使用 scATAC-seq(单细胞染色质可及性测序技术)数据进行训练,以及今年初刚发表于 Nature 的 GET(general expression transformer)模型[22]。同样使用 scATAC-seq 与序列信息,就能在包括此前未见过细胞类型在内的各种情形下,以实验级精度预测基因表达。

此外,还有围绕 Foundation Model 所进行的一些工作,比如说构建检验各种模型模型的基准数据集,基准测试。当然,此方向也是很多人所诟病的“刷指标”而没有实际应用价值的一块。这种批评并非毫无道理,很多指标都直接继承于机器学习,其原设计旨在体现模型预测的精确程度,但并未考虑在不同模型和不同任务上所内含的科学意义,因此确定模型是否真正有效还是需要后续进行湿实验验证,但不乏一些发表于子刊的工作实际都缺少这一点。
在SDbioinfo第十二期《生物大模型的前世今生》播客分享会[23]中,钟博子涛博士就提到:
“仅以生物大模型来说,它能够去做的几乎所有事情都是可被替代的,而不是不可替代。到现在为止,它还很难像结构预测模型那样达到它的不可替代性。”
因为尽管通过掩码建模方式使模型从序列学到了大量的演化信息,但面对 de novo design这样一些零样本任务中,如de novo酶设计,生物模型的表现并不出众,甚至可以说全错。NLP 领域的 Scale定律也在生物模型上部分失效,在一些生物任务中,实测并不是最大的模型效果最好,反而是中等规模的模型表现最佳。显然,尽管生物序列与自然语言有部分共性,尽管测序技术的发展积累了大量生物数据,尽管Transformer算法很好地学到了序列中的演化信息,但与NLP模型不同的是,生物问题无法仅通过数据驱动的方式进行求解,我们需客观、冷静地看待生物模型的发展。
此外,在SDbioinfo第十三期播客分享会[24]中 GET 的共一作者傅熙博士对此工作进行了分享,会中傅博说了一个很有意思的观点:
“AlphaFold之所以影响巨大是因为蛋白质折叠问题通过湿实验解析的成本极大,而在组学领域,技术迅速进步让测序成本降的很低,因此由组学数据训练的模型其更重要的可能反而是通过模型的结果来解析未知的机制。”
在传统的各种组学分析技术之后,至今积累的大量生物学数据又有了一个新的分析方式,由 AI 对数据建模进行拟合,再通过从AI获得的结果与现实进行对比来发现新的认识。
以上,Foundation Model 是一环,但现在更多的且更有应用价值的仍然是上文第一个方向所说对一个 Science 问题中的某个环节建模来替代湿实验或人工。比如药物设计中的蛋白互作、抗体设计、酶工程的酶设计,再到医疗系统内AI病历,AI 解释检测报告,AI 医生,以及阿里达摩院提出已经投入使用的平扫 CT 胃癌筛查模型 DAMO GRAPE[25]等,研究团队在两家医院的模拟机会性筛查试验中,DAMO GRAPE 模型胃癌检出率分别达到 24.5% 与 17.7%,且检出病例中约四成为无症状胃癌患者。一些药企正在逐步尝试用AI优化一些药物发现/设计的环节来提升效率。
方向三
以Science的问题和数据为基础,改进现有的或者构建新的AI算法,最后落点还在Science
需要强调这一方向和 Biology For AI 的区别,B4AI 应该是说借生物学原理构建新的可用于各领域的AI算法,从某种方面来看神经网络或者注意力机制本身就可以部分看作是B4AI的产物。回归原题,上海AI Lab的一个工作在Genome Model上对tokenize方式的进行改进,提出了自动学习DNA序列tokenization策略的MxDNA[26],就是一个完全符合标题的例子:

但这仍只是在现有框架(NLP路径)内的修改。而更有价值且未来我更希望出现的是,从对NLP方向成果的路径依赖走出去,根据各学科数据的特征特点以及所需要解决的问题,设计新的模型架构,从注意力机制到Transformer虽然已经在NLP之外,在各个学科展现了很大的潜力和效果,并且自然语言与生物序列也有一些共通之处,但像Transformer来说其为NLP所设计的本质是不变的。
对于DNA这样极长,有远程作用,重复多,有进化关系,有物种差异,信息分布不均,有特殊\固定位点,有动态的作用机制等等特征的数据,相信会有更适合的算法来对其进行表征。
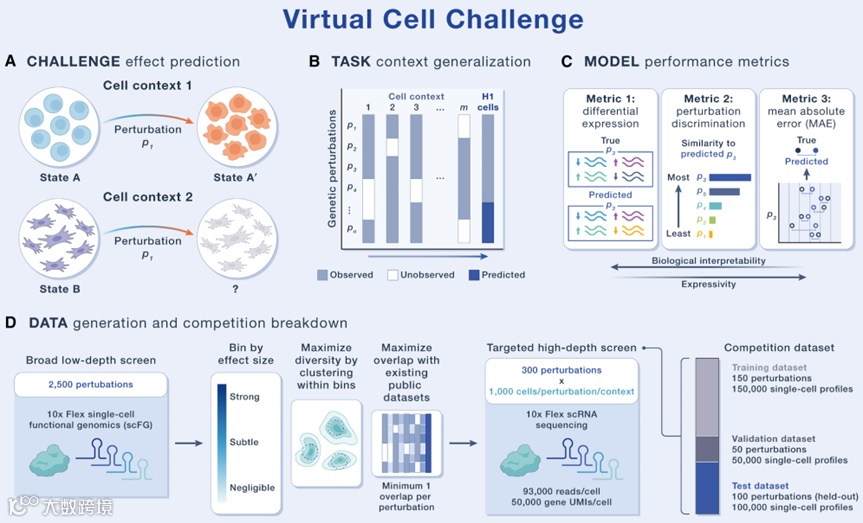
Overview of the Virtual Cell Challenge[27]
今年六月Arc研究所在Cell上发表了题为《Virtual Cell Challenge: Toward a Turing test for the virtual cell》[27]的评论文章,正式推出了虚拟细胞挑战赛。该挑战赛的核心目标是提供一个公平、开放的测试评估,旨在发掘能够通过严格测试的最佳虚拟细胞模型。
据我从参加此赛事的其中一个团队成员所了解,他们初期通过魔改 STATE 模型结果得到了一些提升,但后续就因为数据质量的问题导致对模型的改进在结果上再无提升。究其原因有两个上限:
一方面是测序技术,Perturb-Seq技术仍有一些局限,根本上从测序方面还无法实现对同一批细胞扰动后结果的检测,总之现有公开数据质量较差;
另一方面就是算法性能,Transformer终究不是专为生物数据和生物学问题而设计。
在有限的数据条件下,真正的竞争出现在了谁能更好地利用扰动预测任务背后的生物学意义来针对性修改模型,即一个AI竞赛最后考察的是对生物学的理解深度。
题外话
1.在 AI4Biology 中数据>算法/算力
上个月 GEN 刚发布了对 2024 年诺贝尔化学奖得主、华盛顿大学蛋白设计研究所(IPD)主任David Baker教授的最新专访[28],Baker教授提到:
“深度学习在结构预测上的成功,很大程度上依赖于 PDB 数据库几十年的积累——这是数万人、数十亿美元的共同成果。接下来,AI 能否在更高层级的生物复杂性上(例如细胞层面)发挥预测能力,仍是一个开放问题。”
“PDB 之所以成功,是因为全世界研究者花了几十年,建立了统一标准、集中共享的数据体系。而现在的生物数据则分散且割裂——每家公司都有自己的格式和私有数据集。深度学习需要海量数据。PDB 中约有 20 万个蛋白结构,每个结构包含成千上万个原子坐标,信息量极大。但大多数生物数据集的信息密度远不及此。未来我们需要百万、千万乃至上亿个高质量、标准化且多样化的数据集。只有这样,AI 才能真正跨越蛋白结构层面,进入分子网络与系统级建模。”

GEN专访
在 AI4Biology 中,改进基础算法和堆算力不再成为主流,数据的重要性逐渐突显,并仅次于问题,课题组里有无数据的产出,有无公共数据库之外的独特数据,是否能拿到大体量、覆盖全面、信息含量高、噪音少的高质量数据,直接成为决定能否做出好工作的重要因素。这是因为深度学习的模型本质就是在拟合训练数据,而只有高质量的数据才能得到高效精确的拟合模型。有时甚至反过来,要从数据上去提出问题。
上交大自然科学研究院洪老师组里做的 Venus 系列模型[29],就是在用尽公共数据的同时,还有自己渠道额外的大量数据来进行训练。他们同步推出了蛋白质序列数据集 Venus-Pod[29],其含有近 90 亿条蛋白质序列,包含数亿个功能标签,是全球数据规模最大、功能批注标签最多的数据集,也是 ESM-C[30]模型训练用的 21 亿蛋白质序列的 4 倍体量,以此 Venus 系列模型在一些新的 Benchmark 上,如 ProteinGym[31],实现了最好的效果。
2.AI Co-Scientist 已具备创新能力
cf-PICIs 是一类特殊的移动遗传元件,其是寄生在细菌基因组中的“寄生虫”,它们自身无法移动,需要劫持辅助性噬菌体的复制和组装系统,才能在细菌间传播。通常情况下,噬菌体的宿主范围非常狭窄,一种噬菌体往往只能感染特定种类的细菌。因此,依赖噬菌体传播的遗传元件也应该被限制在少数几种细菌中。但 Penadés 团队观察到一个奇怪的现象:完全相同的 cf-PICI 序列,却频繁地出现在亲缘关系很远的多种不同细菌物种中。cf-PCCIs 如何跨越如此多细菌物种?这个谜题困扰了 Penadés 团队近十年。

AI recapitulates the experimental discovery of a novel gene transfer mechanism[32]
近期,Penadés 团队在其刚刚发表于 Cell 的文章[32]中通过大量实验终于回答了这一问题,不过在好奇心驱使下团队在得到结果后通过 Google 团队基于 Gemini 2.0 构建的多代理架构 “AI Co-scientist” 进行了一场盲测,在只提供一页背景说明与公开资料,不包含任何实验数据的情况下,要求 AI Co-Scientist 给出五个假说,并按可能性进行排名,看是否与团队所得结论一致。
结果是,AI Co-Scientist 在短短两天时间内就独立给出了,与此团队花费十年时间,且刚刚通过大量实验破解但尚未公开发表的生物学机制完全吻合的答案。并在此基础上提出了一系列启发性的新假说,其中一些已经推动此团队开启新的研究方向。从某种方面来说,AI For Science 已经有往 AI Solo Science 发展的趋势,这一点也值得我们思考。
总结
上述三个方向划分不一定严谨,不同背景出身所做的工作也没有一个明显的界限。
大家应该也能看出来,其实在第1点和第2点中我所说的一些例子其实是有些类似的,都是对一个Science问题中的某个环节建模,所以说这就是一种你中有我,我中有你的关系,没有清晰的边界,最终的落脚点还是上一段所说“AI4S的重点是Science”。所有面对开头的问题我的回答只有一句话:AI4S的重点是Science,是要解决的科学问题。AI4S的工作好不好,就要看选择的问题重要性,和对此问题的认识程度。
对于一些志在 AI4S 的同学来说,曾经大多应该都考虑过“去 Science 为主还是 CS 为主的组”这个问题,上文应该能为大家提供一些见解。以第3点为例,没有对 Biology 深入的认识,很难说能将生物学的机制抽象为一种新的算法或者去设计一种针对性的算法,又或是将AI最佳的用在生物学问题之中,而这些生物学认知是在CS组里很难得到的。
参考文献
1. Jumper, J. et al. Highly accurate protein structure prediction with AlphaFold. Nature596, 583–589 (2021).
2. Abramson, J. et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Naturehttps://doi.org/10.1038/s41586-024-07487-w (2024) doi:10.1038/s41586-024-07487-w.
3. Lin, Z. et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science379, 1123–1130 (2023).
4. Huang, K. et al. Biomni: A general-purpose biomedical AIagent. 2025.05.30.656746Preprint athttps://doi.org/10.1101/2025.05.30.656746 (2025).
5. Wierenga, R. P., Golas, S. M., Ho, W., Coley, C. W. & Esvelt, K. M. PyLabRobot: An open-source, hardware-agnostic interface for liquid-handling robots and accessories. Device1, (2023).
6.Nguyen, E. et al. Sequence modeling and design from molecular to genome scale with evo. Science386, eado9336 (2024).
7.Brixi, G. et al. Genome modeling and design across all domains of life with evo 2.
8.Ji, Y., Zhou, Z., Liu, H. & Davuluri, R. V. DNABERT: Pre-trained bidirectional encoder representations from transformers model for DNA-language in genome. Bioinformatics37, 2112–2120 (2021).
9.Zhou, Z. et al. DNABERT-2: Efficient Foundation Model and Benchmark For Multi-Species Genome. Preprint at http://arxiv.org/abs/2306.15006 (2024).
10.Wu, W. et al. GENERator: A long-context generative genomic foundation model. Preprint at https://doi.org/10.48550/arXiv.2502.07272 (2025).
11. Dalla-Torre, H. et al. Nucleotide transformer: Building and evaluating robust foundation models for human genomics. Nat Methods22, 287–297 (2025).
12. Zou, S. et al. A large-scale foundation model for RNA function and structure prediction. 2024.11.28.625345 Preprint at https://doi.org/10.1101/2024.11.28.625345 (2024).
13.Penić, R. J., Vlašić, T., Huber, R. G., Wan, Y. & Šikić, M. RiNALMo: General-purpose RNA language models can generalize well on structure prediction tasks. Preprint at http://arxiv.org/abs/2403.00043 (2024).
14.Wang, N. et al. Multi-purpose RNA language modelling with motif-aware pretraining and type-guided fine-tuning. Nature Machine Intelligence6, 548–557 (2024).
15.Avsec, Ž. et al. AlphaGenome: Advancing regulatory variant effect prediction with a unified DNA sequence model.
16.Cui, H. et al. scGPT: Toward building a foundation model for single-cell multi-omics using generative AI. Nat Methods21, 1470–1480 (2024).
17.Theodoris, C. V. et al. Transfer learning enables predictions in network biology. Nature618, 616–624 (2023).
18.Hao, M. et al. Large-scale foundation model on single-cell transcriptomics. Nat Methods21, 1481–1491 (2024).
19. Levine, D. et al. Cell2Sentence: Teaching large language models the language of biology. 2023.09.11.557287 Preprint at https://doi.org/10.1101/2023.09.11.557287 (2024).
20.Adduri, A. K. et al. Predicting cellular responses to perturbation across diverse contexts with state.
21.Chen, X. et al. EpiAgent: foundation model for single-cell epigenomics. Nat Methods (2025). https://doi.org/10.1038/s41592-025-02822-z
22.Fu, X. et al. A foundation model of transcription across human cell types. Nature637, 965–973 (2025).
23. SDbioinfo线上分享会(第十二期上海交通大学钟博子韬博士):生大模型的前世今生
24. SDbioinfo线上分享会(第十三期哥伦比亚大学博士生傅熙):跨人类细胞类型的转录基础模型
25.Hu, C. et al. AI-based large-scale screening of gastric cancer from noncontrast CT imaging. Nat Med31, 3011–3019 (2025). https://doi.org/10.1038/s41591-025-03785-6
26.Qiao, L. et al. Model decides how to tokenize: Adaptive DNA sequence tokenization with MxDNA. Preprint at https://doi.org/10.48550/arXiv.2412.13716 (2024).
27.Roohani, Y. H. et al. Virtual cell challenge: Toward a turing test for the virtual cell. Cell188, 3370–3374 (2025).
28.https://www.genengnews.com/topics/artificial-intelligence/ai-in-protein-design-hype-vs-reality-explained-by-david-baker/
29. https://huggingface.co/AI4Protein
30.Thomas Hayes et al. Simulating 500 million years of evolution with a language model.Science387,850-858(2025).DOI:10.1126/science.ads0018
31.Notin, P. et al. ProteinGym: Large-scale benchmarks for protein fitness prediction and design.
32.Penadés, J. R. et al. AI mirrors experimental science to uncover a mechanism of gene transfer crucial to bacterial evolution. Cell 188, 6654-6665.e2 (2025).
作者:康凯
⭐ SDbioinfo-往期回顾 ⭐
【栏目1:播客分享会】
【栏目2:前沿信息早知道】
诺奖给了Treg,如何用单细胞测序寻找它的“主控开关”?
【栏目3:知识解读】
马尔科夫链蒙特卡洛方法mcmc原理
【栏目4:最新招聘】
SDbioinfo嘉宾-张心仪博士组招聘

【?我是谁?】
SDbioinfo,专注「计算生物学」领域的学习智库
我们汇聚了来自哈佛剑桥港大、北清复交等高校科研工作者。我们的愿景:链接领域最强大脑,用数据和分析改变生命。
不管你是:
⭐ 找实习、找工作、出国留学
⭐ 找校友、找搭子、找资源
⭐ 找科研方法、前沿信息、大咖分享
统统可以帮你,加入SDbioinfo,和全球生信人一起成长!
(添加微信:SDbioinfo_2023)



