今天要模仿的图片来自于论文 Core gut microbial communities are maintained by beneficial interactions and strain variability in fish。期刊是 Nature microbiology

重复的图片是Figure1中的堆积柱形图和箱线图,然后将其组合
需要的工具及准备
本次可视化基于windows 10系统。
-
数据存储和准备:Microsoft Excel,text
-
数据可视化:R version 4.0.3 和 R Studio,用来要绘制、修饰和整合:堆积柱状图(Stacked Histogram ), 箱图(box plot),柱状图(bar chart),Venn图(Venn diagram),泡泡图(bubble plot),曲线图(Line Graph) 安装请参考:https://www.jianshu.com/p/1a0f25086e8b两者下载地址分别是:R: https://www.r-project.org/R Studio: https://rstudio.com/products/rstudio/download/
-
辅助工具Adobe Photoshop CS4: 做出来的图中的一些文字标记进行一定的修补等。
数据准备与实现:
堆积柱状图(Stacked Histogram ):
-
数据准备:一般可视化最重要的一部分就是数据的准备,请看以下图,给出了对应的数据内容,以下简作说明:第一列:是分组,即共四个组。第二列:每个组里的不同物种。第三列:每个分组里的每个物种的相对丰度。
这样分清层次后,我就自己生成了以下数据,并存为CSV格式。
-
需要的R 包即具体实现过程:
install.packages('ggplot2') #安装ggplot画图包
library(ggplot2)#导入ggplot包
然后就是要读取数据了,为了方便初学者,我用file.choose跳出框来读取:
df<-read.csv(file.choose())
点击enter,会跳出框,选择刚才的csv文件,即完成了读取,数据内容可以点击
df
查看:
说明没问题,是得到了dataframe;这样:数据准备好了,作图的工具也准备好了,那么我们就开始作图:
ggplot(df,aes(x=Status,y=Relative.abundance....,fill=Taxonomy)) + geom_bar(stat = 'identity', width = 0.3, position = 'fill')

说明:ggplot(作图用的dataframe,aes(x=组名的列,y=丰度数值,fill=按照物种类型着色)) + geom_bar(stat = 'identity', width =柱子的宽度 , position = 'fill' 则会铺满整个高度,如果删除则会显示如下这种图(复制以下代码再运行就会明白))
ggplot(df,aes(x=Status,y=Relative.abundance....,fill=Taxonomy)) + geom_bar(stat = 'identity', width = 0.5)

然而发现:背景颜色为灰色而且带着线条,难看,所以去掉背景颜色和线条,即
ggplot(df,aes(x=Status,y=Relative.abundance....,fill=Taxonomy)) + geom_bar(stat = 'identity', width = 0.3, position = 'fill')+theme_set(theme_bw())+theme(panel.grid.major=element_line(colour=NA))
说明:theme_set(theme_bw())#去掉背景色, theme(panel.grid.major=element_line(colour=NA)) #去掉线条颜色
这样就得到以下图,离目标越来越近了:
发现目标图里的横坐标标签存在一定角度的,那么横坐标和纵坐标标签用以下代码实现:
ggplot(df,aes(x=Status,y=Relative.abundance....,fill=Taxonomy)) + geom_bar(stat = 'identity', width = 0.3, position = 'fill')+theme_set(theme_bw())+theme(panel.grid.major=element_line(colour=NA))
+theme(axis.text.x = element_text(face="bold", color="black", size=10, angle=90), axis.text.y = element_text(face="bold", color="black", size=10))

删除横坐标轴的名称:
ggplot(df,aes(x=Status,y=Relative.abundance....,fill=Taxonomy)) + geom_bar(stat = 'identity', width = 0.3, position = 'fill')+theme_set(theme_bw())+theme(panel.grid.major=element_line(colour=NA))+theme(axis.text.x = element_text(face="bold", color="black", size=10, angle=90), axis.text.y = element_text(face="bold", color="black", size=10))
+theme(axis.title.x=element_blank())

发现如果角度设置为45的话,横坐标标签与坐标轴交叉,所以用theme的调节来拉开距离,
p0<-ggplot(df,aes(x=Status,y=Relative.abundance....,fill=Taxonomy)) + geom_bar(stat = 'identity', width = 0.3, position = 'fill')+theme_set(theme_bw())+theme(panel.grid.major=element_line(colour=NA))+theme(axis.text.x = element_text(face="bold", color="black", size=10, angle=45), axis.text.y = element_text(face="bold", color="black", size=10))+theme(axis.title.x=element_blank())+ theme(axis.text.x = element_text(angle = 45, hjust = 0.4, size = 10, vjust=0.4))
得到了较好的图: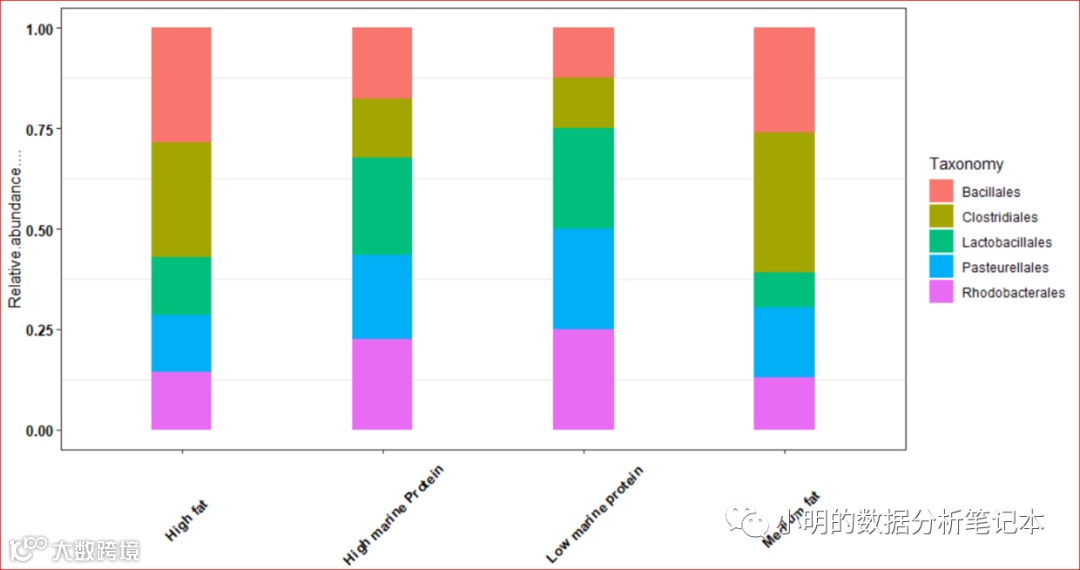
-
如果对其文字格式或字体需要继续调整,可以用ps实现。具体如下:1)首先将上述图输出:  2)用Adobe Photoshop CS4 打开,并选择工具---选择--delete删除文字部分;
2)用Adobe Photoshop CS4 打开,并选择工具---选择--delete删除文字部分; 3)ctrl+shift+N 新建图层--用文字工具输入标签---再点击选择工具,点击文字--输入-45度--应用---得到比较好看的理想图:
3)ctrl+shift+N 新建图层--用文字工具输入标签---再点击选择工具,点击文字--输入-45度--应用---得到比较好看的理想图:
 其他的文字部分也是类似处理并保存:得到下图
其他的文字部分也是类似处理并保存:得到下图
箱图box plot的绘制:
-
简单箱图的绘制 箱图的表示的意义与理解请参考我之前的一个笔记:https://www.jianshu.com/p/54d4996d73cd
箱图数据的格式与要求: 同上:读取数据(是dataframe)
同上:读取数据(是dataframe)
bp<-read.csv(file.choose())
bp
 作图:
作图:
boxplot(bp, col = c("green","brown","purple","blue"),names = c("High marine Protein","Medium fat","High fat","Low marine protein"), ylab = "Richness")
解释:boxplot(bp#选择上面的dataframe, col = c(#给每个箱图选择颜色,用英文标点双引号!"green","brown","purple","blue"),names = c(#给每个箱子命名"High marine Protein","Medium fat","High fat","Low marine protein"), ylab = #给纵轴起名"Richness") 得到: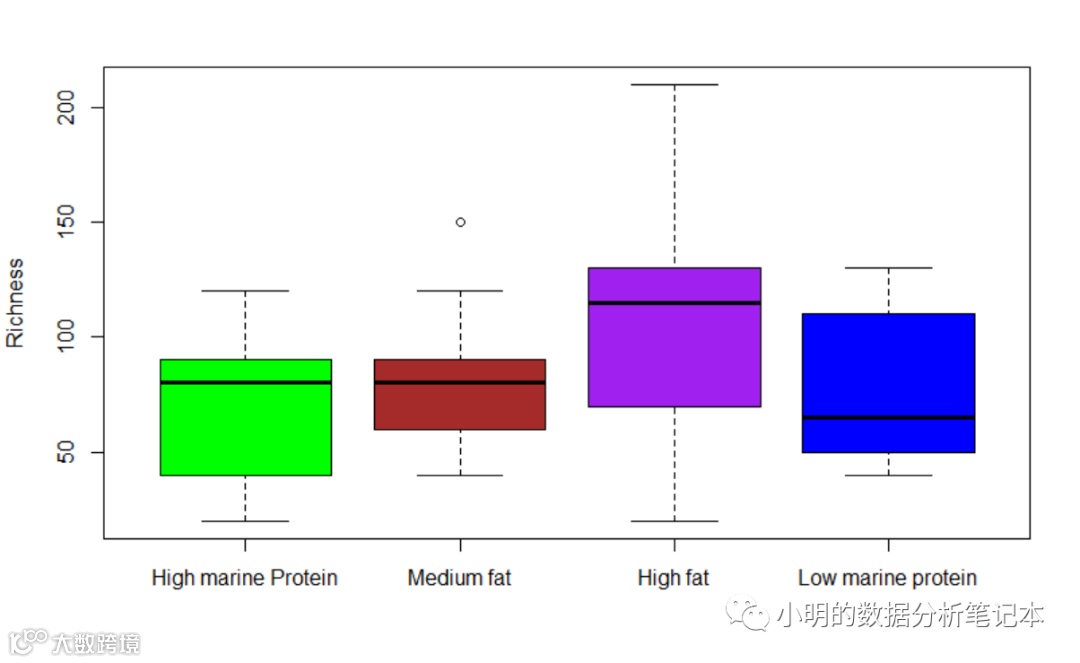
发现目标图中,下标都单独拿出来形成了legend,这是想到其实ggplot直接作box plot就可以直接搞定,但是数据格式略有不同,准备的数据为: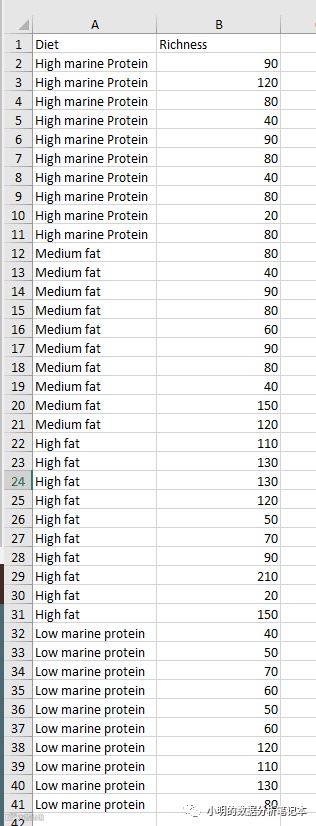
#读取数据
bp<-read.csv(file.choose())
然后进行作图:
ggplot(bp, aes(x=Diet, y=Richness, fill=Diet)) + geom_boxplot()
得到:
这下就按照第一个堆积图的后续修饰,删除横坐标标签:
ggplot(bp, aes(x=Diet, y=Richness, fill=Diet)) + geom_boxplot()+theme(axis.title.x=element_blank())
即得到: 或者再原图基础上可以删除横坐标的所有标签:
或者再原图基础上可以删除横坐标的所有标签:
p1<-p1+theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank())
p1#会看到以下的结果:

第一张箱图先就这么搞定!为了后续合并图形结果,我们把这张赋值为p1, 即:
p1<-ggplot(bp, aes(x=Diet, y=Richness, fill=Diet)) + geom_boxplot()+theme(axis.title.x=element_blank())

箱图2: 带p value的箱图的绘制
先要安装另外两个包:ggsignif这个显著性检验的包
install.packages('ggsignif')
和ggthemr包:
install.packages('ggthemr') #主题配置包
如果ggthemr报错,则:用devtools搞定!方法:
install.packages("devtools")# 先安装或更新
devtools::install_github('Mikata-Project/ggthemr')#安装ggthemr,如果提示选择None
并导入包:
library(ggplot2)
library(ggthemr)
library(ggsignif)
这样就可以做正事了:
#分组
compaired <- list(c("High marine Protein", "Medium fat"), c("High marine Protein","High fat"), c("High marine Protein","Low marine protein"),c("High fat", "Medium fat"),c("High fat", "Low marine protein"),c("High fat", "Low marine protein"))#先进行比较的分组
然后作图:
ggthemr("flat")
p2 <- ggplot(bp, aes(Diet, Richness, fill = Diet)) + geom_boxplot() + geom_signif(comparisons = compaired, step_increase = 0.3, map_signif_level = F, test = wilcox.test)
p2
得到wilcox.test()比较两组(非参数)差异分析标注的箱图,如下: 注:map_signif_level 如果为TRUE,显示方法为""=0.001, ""=0.01, ""=0.05,如果选择f会显示数值 即下图:NS 表示not significant 不显著
注:map_signif_level 如果为TRUE,显示方法为""=0.001, ""=0.01, ""=0.05,如果选择f会显示数值 即下图:NS 表示not significant 不显著 各类检验适用 的情况如下:
各类检验适用 的情况如下: ggsignif主要的一个函数是geom_signif(),使用方法和ggplot2中其他的geom_***()一样,作为图层添加到图形中就可以,他的主要参数为:
ggsignif主要的一个函数是geom_signif(),使用方法和ggplot2中其他的geom_***()一样,作为图层添加到图形中就可以,他的主要参数为:
另外要注意的是:做显著性检验的前提是数据要符合正态分布,对应的正态分布的检验可参考我之前的简单笔记:https://www.jianshu.com/p/0150a9233809
分组箱图的绘制
发现有一个箱图按照组别分箱,所以我们给原数据进行分组,数据如下:
然后对上述结果进行一个分组:
p3<-p2+geom_boxplot()+facet_wrap(~gender)#分箱处理
运行得到分组的箱图:
用ggpubr实现多个箱图的合并与组合:
install.packages('ggpubr')#安装包
library(ggpubr)#加载包
然后给定参数,进行组合:
ggarrange(p0,p1,p2,p3,labels = c("A","B","C","D",ncol=2,nrow=2))#两行两列
说明:输入每个箱图的名字p0,p1,p2,p3,给定图名,几行几列,运行就可以得到组合图:
重要:去掉横坐标标签 对四张图都去除横坐标的标签:用的代码是:
+theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank())
具体实现为
p0<-p0+theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank())
p1<-p1+theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank())
p2<-p2+theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank())
p3<-p3+theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank())
合并的代码
ggarrange(p0,p1,p2,p3,labels = c("A","B","C","D",ncol=2,nrow=2))#两行两列
去除横坐标再合并的结果是:
对于文字部分的修订,文字格式的调整可以参考上述3.1.3 Adobe Photoshop CS4进行美化。
欢迎大家关注我的公众号
小明的数据分析笔记本



