欢迎关注R语言数据分析指南
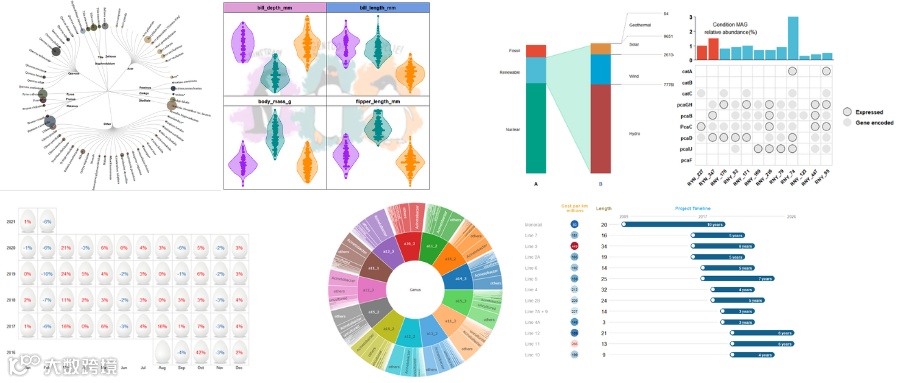
❝本节来解决「npj biofilms and microbiomes」中的一张论文图的绘制问题,「下方代码不会对图形进行复现,旨在介绍一下如何根据已有分组信息来添加背景条带」。下面小编通过一个案例来进行展示,图形仅供展示用过程仅供参考,希望各位观众老爷能够喜欢。数据代码已经整合上传到2023VIP交流群,加群的观众老爷可自行下载,有需要的朋友可关注文末介绍加入VIP交流群
❞
论文

论文原图

图形解读
此图使用门水平的信息进行绘制
-
1.根据OTU丰度表+注释表可以获得每一个门水平的包含的OTU信息 -
2.根据需要在门水平下选取一定量的OTU编号 -
3.进化树的构建则根据选取的OTU编号从代表序列中提取,对代表序列进行多序列比对后构树。
❝完成上述操作后就得到了基础的树图,但是由于我们是根据已有的分组信息来对OTU添加背景条带,此时若是使用「geom_strip」函数来添加条带则会比较麻烦,因此通过循环来添加则比较便捷。
❞
加载R包
library(tidyverse)
library(treeio)
library(ape)
library(magrittr)
library(ggtree)
数据清洗
otu <- read.delim('otu.xls',row.names=1) %>%
select_if(is.numeric) %>% rownames_to_column(var="OTU") %>%
left_join(.,read_tsv("otu.xls") %>% select_if(~!is.numeric(.)),
by="OTU") %>%
separate(taxonomy,
into=c("domain","phylum","class","order","family","genus","species"),sep=";") %>%
mutate_at(vars(c(`domain`:`species`)),~str_split(.,"__",simplify=TRUE)[,2]) %>%
column_to_rownames("OTU") %>%
select(where(is.numeric),phylum) %>% head(200)
聚类分析
tree <- hclust(dist(otu %>% select(where(is.numeric)),method="canberra"))
自定义函数
# 定义函数用于绘制条带并返回绘图对象
draw_strips <- function(p, labels, color) {
for (label in labels) {
p <- p + geom_strip(label, label, extend = 0.5, color = color,
offset = 2.1, barsize = 22, alpha = 0.5)
}
return(p)
}
获取门水平信息
df <- otu %>% rownames_to_column(var="ASV") %>% select(ASV,phylum)
df %>% pull(phylum) %>% unique()
提取ASV-id
# 使用 filter 和 pull 从 df 中提取标签
labels_to_group <- df %>% filter(phylum == "Proteobacteria") %>% pull(ASV)
labels_to_group2 <- df %>% filter(phylum == "Gemmatimonadetes") %>% pull(ASV)
labels_to_group3 <- df %>% filter(phylum == "Actinobacteria") %>% pull(ASV)
labels_to_group4 <- df %>% filter(phylum == "Chloroflexi") %>% pull(ASV)
labels_to_group5 <- df %>% filter(phylum == "Acidobacteria") %>% pull(ASV)
labels_to_group6 <- df %>% filter(phylum == "Rokubacteria") %>% pull(ASV)
绘制树干
p <- ggtree(tree, layout = "circular", branch.length = "none")
绘制不同组的条带
p <- draw_strips(p, labels_to_group, "#4DBBD5FF")
p <- draw_strips(p, labels_to_group2, "#00A087FF")
p <- draw_strips(p, labels_to_group3, "#F39B7FFF")
添加文本
p +
geom_strip("ASV_101653","ASV_56052",extend=0.5,color="yellow",
offset = 2.1, barsize = 22, alpha = 0.5)+
geom_tiplab(size = 2, color = "black",offset = 3)

❝本节内容介绍到此结束,过程仅供参考;有需要学习个性化数据可视化的朋友,欢迎到小编的「淘宝店铺」 「R语言数据分析指南」购买「2023年度会员文档」同步更新中「售价149元」,内容主要包括各种「高分论文的图表分析复现以及一些个性化图表的绘制」均包含数据+代码;按照往年数据小编年产出约在150+以上
❞
购买后微信发小编订单截图即邀请进新的会员交流群,小编的文档为按年售卖,只包含当年度的「除系列课程外」的文档,有需要往年文档的朋友也可下单购买,需要了解更多信息的朋友欢迎交流咨询。
淘宝扫一扫

2023会员群案例展示