欢迎关注R语言数据分析指南
❝本节来分享Nature Genetics一篇泛基因组相关的论文。作者提供了对应的数据及分析代码,此次来简单绘制一个图作为引子,与原文有所不同,个人观点仅供参考。有需要学习R语言绘图的朋友可关注文末介绍购买小编的R绘图文档。购买前请咨询,零基础不要买。
论文信息
A pangenome of maize provides genetic insights into drought resistance Yang, S., Wang, Y., Huang, Q. et al. A pangenome of maize provides genetic insights into drought resistance. Nat Genet (2025). https://doi.org/10.1038/s41588-025-02378-w
Yang, S., Wang, Y., Huang, Q. et al. A pangenome of maize provides genetic insights into drought resistance. Nat Genet (2025). https://doi.org/10.1038/s41588-025-02378-w
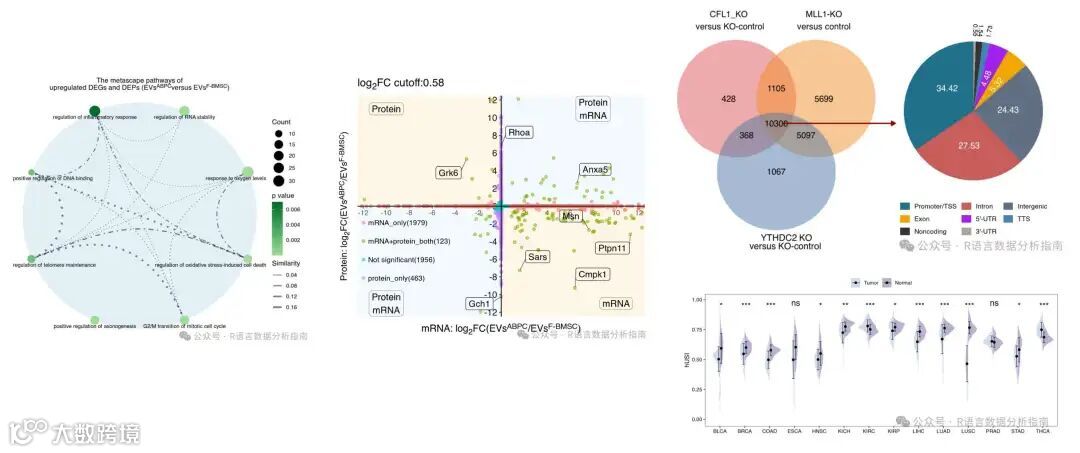
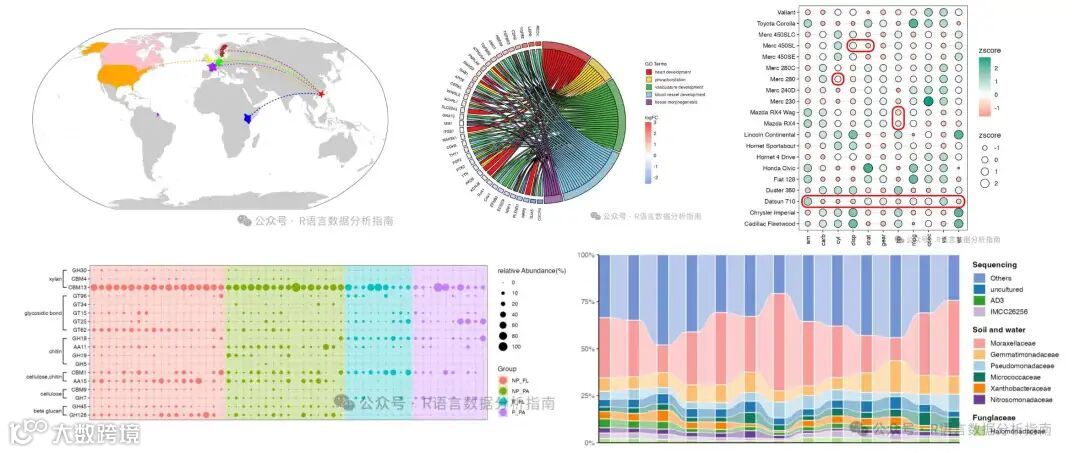
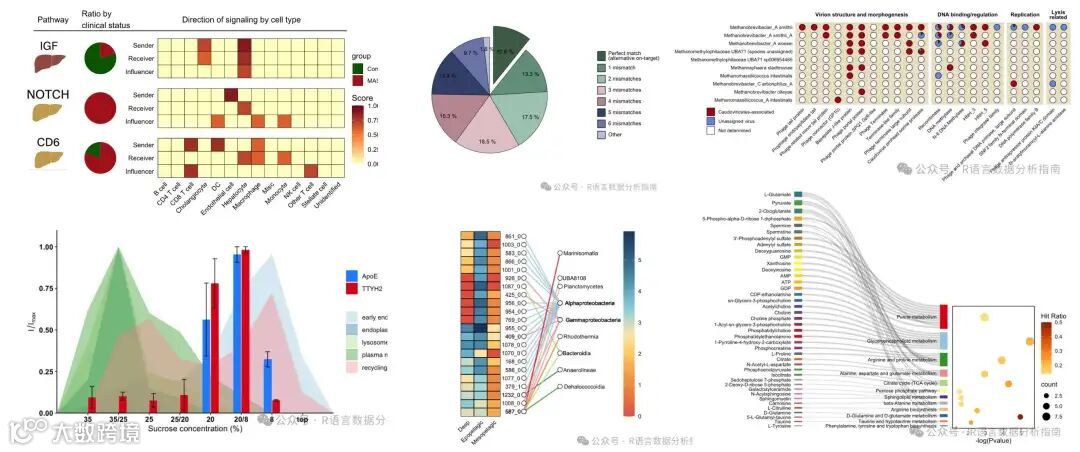
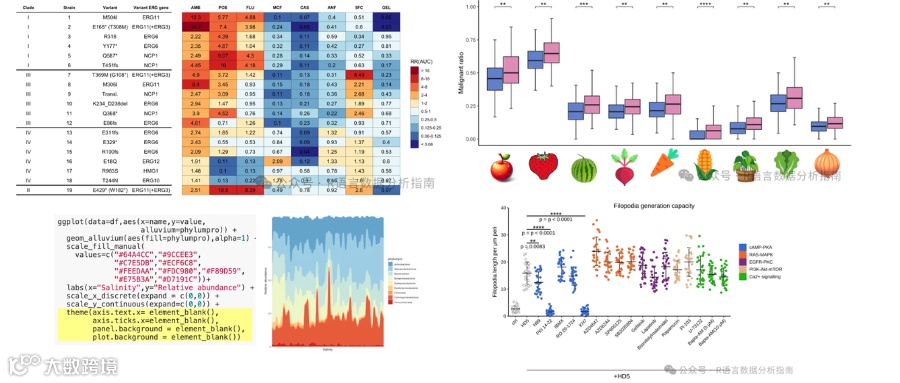
论文图

代码展示
library(tidyverse)
library(patchwork)
# 条图
df <- read_tsv("data.tsv")
p1 <- ggplot(data=df,aes(factor(`Present genome number`),`Gene family number`)) +
geom_col(aes(fill=Group)) +
scale_fill_manual(values = c(
Core = "#E41A1C",Softcore = "#FB9A06",
Dispensable = "#4DBBD5",Singletons = "#984EA3")) +
scale_x_discrete(breaks=seq(0,56,by=4)) +
scale_y_continuous(limits = c(0,20000),expand = c(0,0)) +
labs(x="Present genome number",y="Gene family number") +
theme_classic() +
theme(axis.text=element_text(color="black"),
legend.position = "none")
饼图
df2 <- read_tsv("pie.tsv") %>%
mutate(group = sub(" .*", "", Type)) %>%
filter(Type!="Singletons")
df2$percentage <- df2$Number / sum(df2$Number) * 100
p2 <- ggplot(data=df2,aes(x="",y=Number,fill=group)) +
geom_bar(width = 1,stat = "identity") +
geom_text(aes(label =paste0(round(percentage,1),"%")),
position = position_stack(vjust = 0.5))+ # 添加标签
scale_fill_manual(values = c(
Core = "#E41A1C",Softcore = "#FB9A06",
Dispensable = "#4DBBD5",Private = "#984EA3")) + coord_polar("y") +
theme_void()+
theme(legend.title = element_blank())
拼图
inset_element() 通过控制(right - left)和(top - bottom)的差值来控制嵌入图形的大小。
p1 + inset_element(p2,
left = 0.2, right = 0.9,
bottom = 0.3, top = 1)

关注下方公众号下回更新不迷路
购买介绍
❝本节介绍到此结束,有需要学习R数据可视化的读者欢迎到淘宝店铺:R语言数据分析指南,购买小编的R语言可视化文档,2025年购买将获取2025年更新的绘图内容,同时将赠送2024年的绘图文档内容,其余内容无。
更新的绘图内容包含数据+代码+注释文档+文档清单,小编只分享案例文档,不额外回答问题,无答疑服务,更新截止2025年12月31日结束,后续不在进行任何更新,零基础基础一般不推荐买。
在线目录大纲

淘宝店铺

2025年更新案例图展示